- 2022-08-18 发布 |
- 37.5 KB |
- 85页


申明敬告: 本站不保证该用户上传的文档完整性,不预览、不比对内容而直接下载产生的反悔问题本站不予受理。
文档介绍
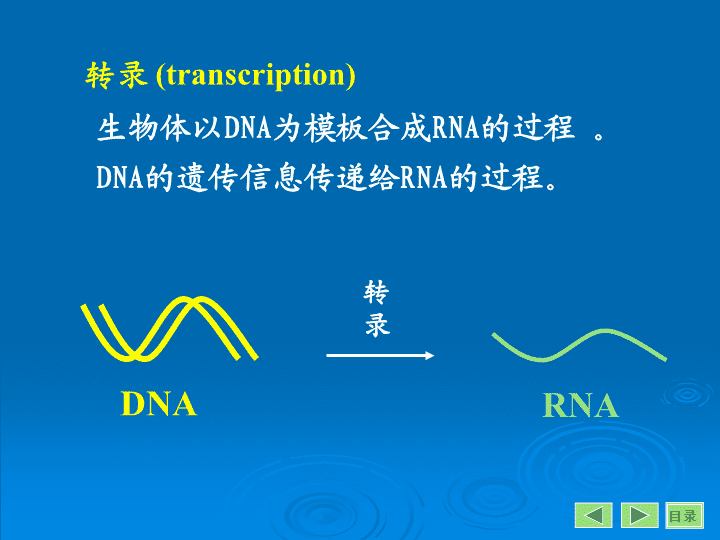
(农学)生物化学课件 转录
第十一章RNA的生物合成(转录)RNABiosynthesis,Transcription\n转录(transcription)生物体以DNA为模板合成RNA的过程。DNA的遗传信息传递给RNA的过程。转录RNADNA\n复制和转录的共同点:DNA模板依赖DNA的聚合酶碱基配对规律生成磷酸二酯键链延长方向5'→3'\n复制和转录的区别\n模板和酶TemplatesandEnzymes第一节\n5′···GCAGTACATGTC···3′3′···cgtgatgtacag···5′5′···GCAGUACAUGUC···3′N······Ala·Val·His·Val······C编码链模板链mRNA蛋白质转录翻译一、转录模板结构基因(structuralgene):能作为转录模板的DNA区段。\n模板链(templatestrand)有意义链或Watson链DNA双链中,能按碱基配对规律指引转录,生成RNA的一股单链。编码链(codingstrand)反义链或Crick链DNA双链中碱基序列与RNA一致的一股链。反义核酸:以编码链为模板合成的核酸,称为反义核酸(反义RNA、反义DNA)。序列与模板链一致,能抑制mRNA表达。\n5335模板链编码链编码链模板链结构基因转录方向转录方向不对称转录(asymmetrictranscription)在DNA分子双链上某一区段,一股链用作模板指引转录,另一股链不转录。模板链并非永远在同一条单链上。\n二、RNA聚合酶定义:以DNA为模板,催化三磷酸核苷(NTP)聚合形成RNA的酶。作用特点:DNA为模板不需引物,两游离NTP或一游离NTP与RNA链聚合.3'-OH与5'-P聚合形成磷酸二酯键。方向:5'→3'。\n(一)原核生物的RNA聚合酶\n核心酶(coreenzyme)全酶(holoenzyme)核心酶:转录延长;全酶:转录起始。σ70是辨认典型转录起始点的蛋白质。σ32是辨认热休克蛋白(Hsp)转录起始点的蛋白质。\nRNA聚合酶全酶在转录起始区的结合\n(二)真核生物的RNA聚合酶45S-rRNA,5S-rRNA:核糖体RNA成分。hnRNA:mRNA的前体。snRNA:参与mRNA剪切。\nRNA聚合酶结构特点:两个大亚基:分子量>100KDa,催化亚基,与原核β,β'亚基有同源性。50kDa亚基:分子量50kDa,与原核α亚基相似。小亚基:10,7,11个。羧基末端结构域(CTD):(YSPTSPS)n:Y:Tyr;S:Ser;P:Pro;T:ThrRNA-polⅡ大亚基C末端磷酸化,使转录进入链的延长阶段。\n三、模板与酶的辨认、结合操纵子(operon):原核生物的一个转录单位,包括若干个结构基因及其上游(upstream)的调控序列。5335结构基因调控序列RNA-pol启动子(promoter):RNA聚合酶结合模板DNA的部位。\nRNA聚合酶保护法目录\n开始转录TTGACAAACTGT-35区(Pribnowbox)TATAATPuATATTAPy-10区1-30-5010-10-40-205335原核生物启动子保守序列RNA-pol辨认位点(recognitionsite)55RNA聚合酶保护区结构基因33\n\n原核生物启动子结构特点:一致性序列(concensus保守序列):碱基序列相对稳定,不易发生突变。TTGACA:-35区,RNA聚合酶的辨认位点,σ亚基结合位点。TATAAT:-10区,Pribrow盒,RNA聚合酶的稳定结合位点。β'亚基的结合位点。\nTATA盒CAAT盒GC盒增强子顺式作用元件结构基因-GCGC---CAAT---TATA转录起始真核生物启动子保守序列\n转录过程TheProcessofTranscription第二节\n一、原核生物的转录过程\n(一)转录起始转录起始需解决两个问题:RNA聚合酶必须准确地结合在转录模板的起始区域。DNA双链解开,使其中的一条链作为转录的模板。\n辨认起始位点:亚基辨认启动子的-35区,RNA聚合酶全酶(2)与模板疏松结合。酶滑行到-10区,通过亚基与模板牢固结合。2.DNA双链解开:RNA聚合酶挤入DNA双链中,解链长度约20个核苷酸,拓扑异构酶参与。转录起始过程:\n3.在RNA聚合酶作用下发生第一次聚合反应,形成转录起始复合物。RNApol(2)-DNA-5'pppGpN-OH3转录起始复合物:RNA聚合酶-DNA模板-四磷酸二核苷酸5-pppG-OH+NTP5-pppGpN-OH3+ppi\n(二)转录延长1.亚基脱落,RNA–pol聚合酶核心酶变构,与模板结合松弛,沿着DNA模板前移;2.在核心酶作用下,NTP不断聚合,RNA链不断延长。(NMP)n+NTP(NMP)n+1+PPi3.转录空泡的形成:\n3.转录空泡(transcriptionbubble):DNA解开的两条单链与RNA聚合酶及其转录产物RNA构成的转录复合物。目录RNA-pol:40bp以上;解链:20bp;RNA/DNA:12bp。\n目录DNA/DNA双链比DNA/RNA杂化双链稳定稳定性:G≡C>A=T>A=UDNA双链超螺旋作用\n53DNA原核生物转录过程中的羽毛状现象核糖体RNARNA聚合酶\n依赖Rho(ρ)因子的转录终止非依赖Rho因子的转录终止(三)转录终止RNA聚合酶在DNA模板上停止前进,转录产物RNA链从转录复合物上脱落。机制\nATP依赖Rho因子的转录终止:具有控制转录终止作用的蛋白质因子。机制:Rho因子与RNA3’-OH的polyC结合,抑制聚合酶活性,激活解螺旋活性。\n2.非依赖Rho因子的转录终止DNA模板近终止处,有特殊的碱基序列,转录出RNA产物形成特殊的结构终止转录。发夹结构:反向碱基互补序列末端PolyU:U:A不稳定.\n5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU...3`5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU...3`RNA5TTGCAGCCTGACAAATCAGGCTGATGGCTGGTGACTTTTTAGTCACCAGCCTTTTT...3DNAUUUU...…UUUU...…5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU...3`茎环(stem-loop)/发夹(hairpin)结构\n茎环结构终止转录的机理使RNA聚合酶变构,转录停顿;末端polyU使转录复合物趋于解离,RNA产物释放。5´pppG5335RNA-pol\n\n二、真核生物的转录起始(一)转录起始转录起始上游区段比原核生物多样化,转录起始时,RNA-pol不直接结合模板的启动子,其起始过程比原核生物复杂。\n转录起始点-25bpTATA盒CAAT盒GC盒增强子顺式作用元件(cis-actingelement):DNA分子上可影响(调控)转录的序列。启动子,增强子,沉默子。1.转录起始前的上游区段:AATAAA切离加尾转录终止点修饰点外显子翻译起始点内含子OCT-1OCT-1:ATTTGCAT八聚体\n2.转录因子(transcriptionalfactors,TF)能与顺式作用原件识别、结合,调节真核基因转录的蛋白质,称为转录调节因子,简称转录因子。反式作用因子(trans-actingfactors)trans-分子外顺式作用元件(cis-actingfactor)cis-分子内\n基本转录因子(TFⅠ,TFⅡ,TFⅢ)直接或间接参与RNA聚合酶与启动子结合的转录因子特异转录因子:特异调节各个基因转录的转录因子,决定该基因的时间,空间特异性表达。\n参与RNA-polⅡ转录的TFⅡTBP:TATAbindingprotein,TATA结合蛋白TAF:TBPassociatedfactor,TBP辅因子,不同基因TAF不同CTD:RNA-polⅡ大亚基羧基末端结构域\n3.转录起始前复合物(pre-initiationcomplex,PIC)真核生物RNA-pol不与DNA分子直接结合,而需依靠众多的转录因子。\nPOL-ⅡTFⅡFⅡAⅡB由RNA-PolⅡ催化转录的PICPOL-ⅡTFⅡFⅡHⅡETBPTAFTFⅡD-ⅡA-ⅡB-DNA复合物TATAⅡAⅡBTBPTAFTATAⅡHⅡECTD-PPIC组装完成,TFⅡH使CTD磷酸化\n(二)转录延长RNA-pol前移,核小体移位和解聚现象。体外转录实验:移位DNA酶水解法,200bp及其倍数的电泳区带,被组蛋白保护。细胞转录实验:解聚组蛋白精氨酸乙酰化,降低正电荷。DNA:AMP→ADP→聚ADP,降低负电荷。与原核生物大致相似,没有转录与翻译同步的现象。\nRNA-PolRNA-PolRNA-Pol核小体转录延长中的核小体移位转录方向\n5------AAUAAA-5------AAUAAA--核酸酶-GUGUGUGRNA-polAATAAAGTGTGTG转录终止的修饰点55333加尾AAAAAAA······3mRNA(三)转录终止——和转录后修饰密切相关。转录终止修饰点:DNA读码框架下游的一组AATAAA和GT共同序列,参与转录终止过程,这些序列称之。\n真核生物的转录后修饰Post-transcriptionalModification第三节\n几种主要的修饰方式1.剪接(splicing)2.剪切(cleavage)3.修饰(modification)4.添加(addition)\n一、真核生物mRNA的转录后加工(一)首、尾的修饰5端形成帽子结构(m7GpppGp—)3端加上多聚腺苷酸尾巴(polyAtail)\n帽子结构:(m7GpppGp—)7-甲基鸟嘌呤-三磷酸鸟苷5’-5’磷酸二酯键\n5pppGp…5GpppGp…pppGppi鸟苷酸转移酶5m7GpppGp…甲基转移酶SAM帽子结构的生成5ppGp…磷酸酶Pi\n帽子结构的功能:核内生成,保护mRNA不被核酸外切酶水解。与翻译过程有关,帽子结构结合蛋白是翻译起始必需的因子。\n2.3’-末端PolyA尾:(长约100-200bp)生成:在核内修饰,与转录终止同时进行.作用:增加mRNA稳定性,维持翻译模板活性。5------AAUAAA-5------AAUAAA--核酸酶-GUGUGUGRNA-polAATAAAGTGTGTG转录终止的修饰点55333加尾AAAAAAA······3mRNA\n鸡卵清蛋白成熟mRNA与DNA杂交电镜图DNAmRNA目录(二)mRNA的剪接\n编码区和非编码区互相间隔,又连续镶嵌而成,这些基因称为断裂基因。1.断裂基因(splitegene)CABD编码区A、B、C、D非编码区\n2.外显子(exon)和内含子(intron)外显子:(基因上的编码序列)在断裂基因及其初级转录产物上出现,并表达为成熟RNA的核酸序列。内含子:(基因上的非编码序列)在断裂基因及其初级转录产物上出现,而在剪接过程中被除去的核酸序列。\n3.内含子的分类:基因的类型和剪接的方式I类:线粒体、叶绿体及某些低等真核生物的rRNA基因;II类:线粒体、叶绿体mRNA基因;III类:自我剪切III类:多数mRNA基因;套索结构剪接IV类:是tRNA基因,剪接过程需酶及ATP。翻译后内含子:胰岛素原,酶原。\nmRNA的剪接过程:Ⅲ类内含子剪接,套索状剪接除去hnRNA中的内含子,将外显子连接。\n鸡卵清蛋白基因hnRNA首、尾修饰hnRNA剪接成熟的mRNA鸡卵清蛋白基因及其转录、转录后修饰目录\n①snRNP剪接体的形成:核内的蛋白质小分子核糖核酸蛋白体(snRNP)(剪接体,splicesome)snRNAsnRNA:(smallnuclearRNA)100-300核苷酸,以U分类:U1-6snRNP:(smallnuclearribonucleoprotein)作为mRNA剪切的场所。\n②内含子两端与U1,U2配对结合。5’剪接点:↓GU--;3’剪接点:--AG↓分支点序列:3’剪接点上游20~50核苷酸范围内保守序列UACUAAC,倒数第二个A为分支点。目录\n③形成完整剪切体,内含子成套索状,外显子靠近。③UACUACA-AGUGU4U5U6E1E2U1U2UACUACA-AGUGU6E1E2U1、U4、U5\npG-OH(ppG-OH,pppG-OH)U-OHGpUpGpA第一次转酯反应第二次转酯反应UpAGpU外显子1内含子外显子2G-OHUpUpGpA④二次转酯反应(twicetransesterification)I类内含子\n剪接体与剪接图示\nhnRNA剪接简图\n5.mRNA的编辑(mRNAediting)mRNA转录后出现核苷酸的替换,插入或缺失,改变DNA模板来源的遗传信息,翻译出氨基酸序列不同的多种蛋白质。\napoBmRNA的编辑apoB基因:29个外显子,14500bp,4536aaapoBmRNA编辑蛋白(REPP,脱氨酶)apoBmRNA第26个外显子第6666位C脱氨→U;密码:CAA(Gln2153)→UAA(终止密码);肝细胞:不表达REPP4536aaapoB100肠上皮细胞:表达REPP2152aaapoB48\n人类apoB基因mRNA(14500个核苷酸)肝脏apoB100(分子量为500000)肠道细胞apoB48(分子量为240000)mRNA编辑\nRNA编辑的生物学意义:产生多种表达产物,产生多种生物学效应,产生功能用途的分化,因此也称为分化加工(differentialRNAprocessing)。DNA上的基因数要比表达的蛋白质种类少。估计10万个基因,实际3万个基因。\n二、tRNA的转录后加工tRNA前体RNApolⅢTGGCNNAGTGCGGTTCGANNCCDNA目录\nRNAaseP、内切酶目录5’前导序列:RNAaseP中部内含子:内切酶\ntRNA核苷酸转移酶、连接酶ATPADP目录\n碱基修饰(2)还原反应如:UDHU(3)核苷内的转位反应如:Uψ(4)脱氨反应如:AI如:AAm(1)甲基化(1)(1)(3)(2)(4)目录\n三、rRNA的转录后加工纵列串联基因:可转录的DNA片段与不能转录的间隔区串联组成的基因。(基因间隔)高度重复序列:间隔区和可转录区可以重复数百个到数千个以上,每重复一次构成一个重复单位,转录一个rRNA,而且每个重复单位转录的rRNA大小基本一致,45SrRNA。丰富基因:具有高度重复序列的基因,称之。rDNA,5srRNA基因,组蛋白基因,免疫球蛋白rRNA基因(rDNA)的特点:\n转录45S-rRNA剪接18S-rRNA5.8S和28S-rRNArDNA内含子内含子28S5.8S18S\n四、核酶具有酶促活性的RNA称为核酶。核酶(ribozyme)\n四膜虫rRNA内含子的二级结构四膜虫rRNA的剪接采用自我剪接(核酶)方式。rRNA前体(35S),内含子(9S)414核苷酸5´-端核苷酸序列\nⅠ类内含子的特殊结构\n\npG-OH(ppG-OH,pppG-OH)U-OHGpUpGpA第一次转酯反应第二次转酯反应UpAGpU外显子1内含子外显子2G-OHUpUpGpA④二次转酯反应(twicetransesterification)I类内含子\n目前研究最多的核酶是小分子RNA核酸内切酶家族:锤头核酶,发夹核酶,丁肝病毒等.\n底物部分核酶的结构特点:通常由60个核苷酸左右组成,底物部分:含有GU序列催化部分:锤头结构,3个茎,1-3个环保守序列:13个碱基\n人工设计的核酶黄线表示合成的核酸分子细线表示天然的核酸分子X表示一致性序列箭头表示切断点目录\n核酶研究的意义核酶的发现(Cech和Altman)1989诺贝尔化学奖;核酶的发现是对传统酶学的挑战;利用核酶的结构设计合成人工核酶。\n澳大利亚Adelaide大学Symons实验室:人工合成一个13寡聚核糖核苷酸一个天然41寡聚核苷酸(类病毒RNA)在体外实验中可以形成锤头结构天然的41寡聚核苷酸水解:9,32个核苷酸两片段\n已合成切割H-ras肿瘤基因的核酶,通过电穿孔的方法导入人黑色素瘤细胞,可以改变肿瘤的表型。建立切割HIV-1病毒RNA核酶的细胞模型。查看更多